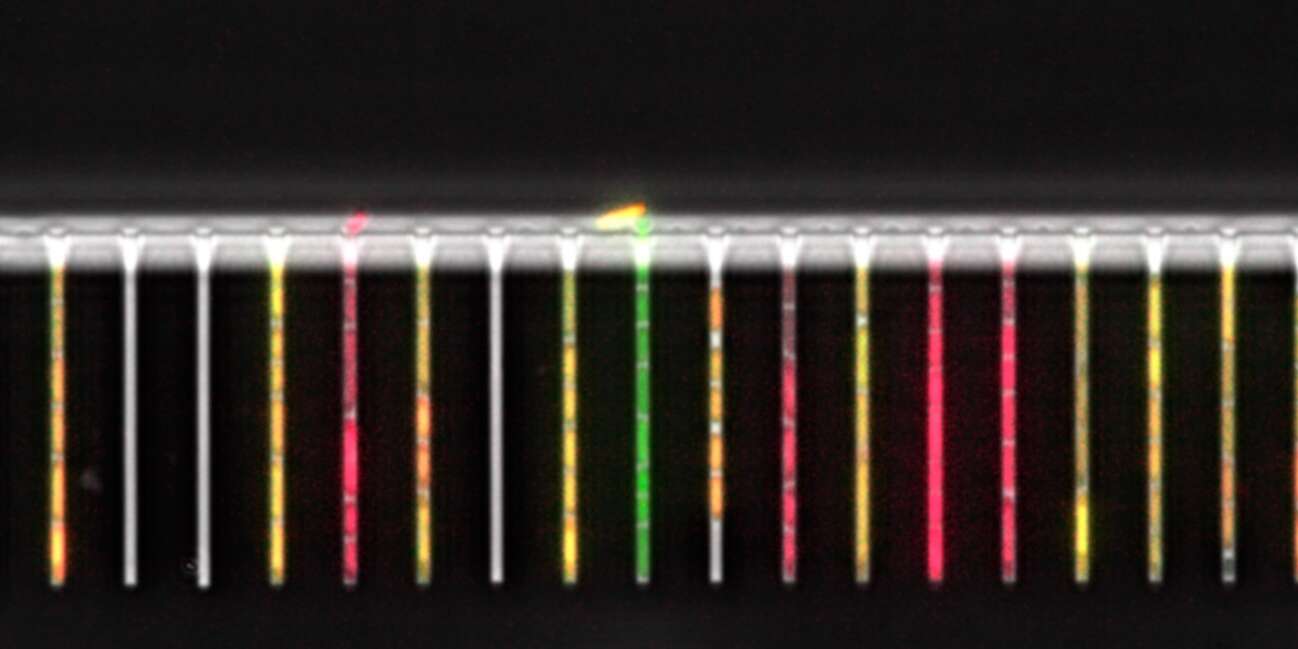
Er ist kaum grösser als eine Streichholzschachtel und doch befindet sich auf diesem Chip ein Labor en miniature. In etwa 2000 Kanälen von einem tausendstel Millimeter Durchmesser wachsen einzelne Bakterienzellen, die die Forscher um Prof. Erik van Nimwegen vom Biozentrum der Universität Basel genau untersuchen können. Denn die Aufnahme von Tausenden von mikroskopischen Bildern in kürzesten Zeitabständen erlaubt es, das Wachstum und Verhalten mehrerer Generationen einzelner E. coli-Bakterien über Tage hinweg zu verfolgen. Die dabei anfallende Fülle an Rohdaten wird durch eine neuartige Software zur Bildauswertung namens MoMA automatisch analysiert und präzise quantifiziert. MoMA wurde gemeinsam mit Wissenschaftlern aus der Gruppe von Prof. Gene Myers am Max-Planck-Institut für Molekulare Zellbiologie und Genetik in Dresden entwickelt.
Mit Mikrofluidik das Verhalten einzelner Zellen analysieren
«Diese Publikation ist das Ergebnis einer mehr als fünf Jahre währenden, sehr intensiven Zusammenarbeit mit dem Labor von Gene Myers. Es war wirklich eine gewaltige Anstrengung, die uns einiges Kopfzerbrechen bereitet hat», sagt Erik van Nimwegen. «Mit unserem Mikrofluidik-Chip und der dazugehörigen Software können wir nun untersuchen, wie Gene in einzelnen Zellen genau reguliert werden, wenn sich die Umweltbedingungen verändern. So erhalten wir nicht nur einen Einblick in genregulatorische Prozesse, sondern auch einen Überblick über die Diversität an Reaktionen mit denen Bakterien auf Umwelteinflüsse antworten.»
So lässt sich beispielsweise untersuchen, wie einzelne Bakterienzellen reagieren, wenn sie einem Antibiotikum ausgesetzt werden, ob sie absterben oder ihr Wachstum einstellen oder sich einfach ungestört weitervermehren und wie die Zellen sich verhalten, je nachdem wie lange das Antibiotikum einwirkt? Für das Verständnis, warum nicht immer alle Krankheitserreger durch Antibiotika getötet werden und Infektionen chronisch werden, ist es wichtig zu wissen, wie sich einzelne Bakterien verhalten. «Mit dem Mikrofluidik-Chip kann man aber auch andere Fragestellungen beantworten, zum Beispiel wie Bakterien miteinander kommunizieren, wie sie auf Stress reagieren oder inwiefern familiäre Beziehungen eine Rolle bei Anpassungsstrategien spielen», so van Nimwegen. «Solche Einzelzellanalysen sehe ich als sehr wichtig an, denn oftmals sind die Messungen von Zellgemeinschaften irreführend, da die Heterogenität der einzelnen Zellen herausgemittelt wird.»
Zelluläres Gedächtnis ist wichtig für schnelle Anpassung
In welchem Masse einzelne Gene reguliert werden, lässt sich mithilfe von leuchtenden Proteinen ermitteln. Je nachdem wie stark das im Fokus stehende Gen aktiviert ist, umso mehr oder weniger dieser fluoreszierenden Proteine werden im Bakterium produziert und die Leuchtstärke variiert dementsprechend. «Wir haben mithilfe des grün fluoreszierenden Proteins beobachten können, wie sich E. coli-Bakterien an einen regelmässigen Wechsel zwischen den Nährstoffen Glukose und Laktose anpassen. Das dazugehörige regulatorische System, das sogenannte Lac-Operon, ist das ‹Wasserstoff-Atom› der Biologie. Es wird seit mehr als fünfzig Jahren erforscht und dennoch konnten wir mit unseren Einzelzell-Analysen ganz neue Eigenschaften aufdecken», sagt van Nimwegen. «So stellen die Bakterien ihren Stoffwechsel im ersten Durchgang mit zeitlicher Verzögerung auf die Laktose-Verwertung um. Wir haben herausgefunden, dass die Bakterien dies auf unterschiedliche Art und Weise tun.» Bei wiederholten Wechsel von Glukose zu Laktose fand dieses Umschalten viel schneller statt, die Zellen fangen viel eher wieder an zu wachsen. Erstaunlicherweise ist die zeitliche Verzögerung der Reaktion bei miteinander verwandten Bakterien sehr ähnlich. Demnach scheinen die Zellen das Verhalten ihrer Vorfahren in Erinnerung zu behalten.
Durch den Einsatz des Mikrofluidik-Systems konnten die Forscher neue Erkenntnisse über die Dynamik der Genregulation und die Anpassungsstrategien von Bakterien gewinnen. Dieses System eignet sich für ein breites Spektrum von Anwendungen. Alle relevanten Informationen zum Chip-Design und den Experimenten, die MoMA-Software zur Bildanalyse sowie die benötigten Originaldaten sind online frei zugänglich.
Originalartikel:
Matthias Kaiser, Florian Jug, Thomas Julou, Siddharth Deshpande, Thomas Pfohl, Olin Silander, Gene Myers, and Erik van Nimwegen. Monitoring single-cell gene regulation under dynamically controllable conditions with integrated microfluidics and software. Nature Communications, published online 15 January 2018
Kontakt: Kommunikation, Katrin Bühler